LINEA DI RICERCA
DESTINO AMBIENTALE
Valutazione degli effetti antibiotici sulle comunità microbiche naturali
Gli effetti antibiotici sulle comunità microbiche naturali
L’utilizzazione agronomica degli effluenti zootecnici e l´impiego a scopi energetici delle biomasse sono pratiche sempre più diffuse nelle aziende agro-alimentari. Gli agroecosistemi richiedono un notevole impiego di energia sussidiaria (carburanti per le macchine agricole, fertilizzanti, pesticidi, irrigazione, mangimi) e non sono più in grado di riciclare la sostanza organica a causa dell’alterazione dei cicli degli elementi dovuta all’intervento continuo dell’uomo (es. rimozione della biomassa prodotta nelle colture o eccesso di accumulo di sostanza organica negli allevamenti) con notevoli impatti sulla qualità del suolo agrario.
In effetti tra i fenomeni più diffusi di degrado del suolo vi sono la perdita di sostanza organica nelle colture e l’eccessivo accumulo di scarti, sotto forma per esempio di letame, negli allevamenti di bestiame. In questi ultimi vengono anche utilizzati grandi quantità di antibiotici per curare e prevenire la diffusione di infezioni. Al fine di smaltire gli scarti organici, in un contesto di economia circolare, molte aziende agricole si avvalgono dell’utilizzo di letame come ammendante del suolo o più recentemente per alimentare impianti di digestione anaerobica da cui ottenere energia sotto forma di biogas. Il biogas prodotto (ad elevato contenuto in metano > 50%) è utilizzato all’interno dell’azienda per produrre energia elettrica e calore, riducendo pertanto il consumo di carburanti fossili e contribuendo alla ciclizzazione dell’anidride carbonica. Oltre al biogas, dal processo di digestione anaerobica si ottiene anche digestato che può essere utilizzato come fertilizzante organico. Poiché sia il letame che il digestato derivano dalle deiezioni animali queste possono contenere residui di antibiotici che non vengono del tutto metabolizzati dagli organismi trattati. Gli antibiotici sono contaminanti emergenti, cioè sostanze non incluse nella legislazione attuale europea e per le quali pertanto non sono stati fissati dei valori soglia di riferimento né nelle acque, né tantomeno nei suoli. Tuttavia è ormai evidente che la loro presenza sia molto diffusa nell’ambiente e che la contaminazione da antibiotici sia la principale causa della diffusione tra ecosistemi e uomo e viceversa del fenomeno dell’antibiotico resistenza.
Focus sull’attività
Obiettivi
La linea di attività 1 ha come obiettivo quello di valutare il destino ambientale degli eventuali residui di antibiotici e relativi geni della resistenza presenti nel letame o nel digestato quando questi vengono utilizzati come ammendanti organici del suolo.
Metodi
Attraverso l’utilizzo di microcosmi sperimentali i campioni di suolo sono stati ammendati con letame o digestato e il destino ambientale residui di antibiotici e relativi geni della resistenza è stato studiato.
Risultati
Gli esperimenti condotti in microcosmi di suolo, che sono stati ammendati o con letame o con digestato in presenza/assenza degli antibiotici, hanno fornito risultati interessanti.
L’aggiunta del digestato al suolo favorisce una comunità microbica più ricca (maggiore biodiversità) e un minore contenuto di batteri patogeni rispetto a quanto osservato quando viene aggiunto il letame. In particolare, la presenza di antibiotici influenza negativamente solo nella fase iniziale l’abbondanza di alcuni gruppi microbici nel suolo ammendato con il digestato. Gli antibiotici studiati vengono degradati nel suolo, sebbene i fluorochinoloni si confermano sempre più persistenti dei sulfonamidi. Diversamente nel suolo in cui è stato aggiunto il letame, si osserva una maggiore persistenza di batteri patogeni e/o portatori di geni di resistenza agli antibiotici.
Ulteriori studi sono in corso per verificare se sullo stesso terreno e con gli stessi ammendanti la crescita di specie vegetali edibili possa influenzare la degradazione degli antibiotici, la trasmissione dell’antibiotico resistenza ed eventualmente bioconcentrare sia gli antibiotici, che gli stessi geni. Questo ultimo aspetto è particolarmente rilevante per valutare una possibile trasmissione di tali contaminanti emergenti dall’ambiente all’uomo tramite l’assunzione di alimenti vegetali crudi.
Attività sperimentale in microcosmi di laboratorio
Campioni di suolo trattati con letame bovino o digestato e in alcuni casi anche con gli antibiotici oggetto di studio (sulfametossazolo, ciprofloxacina, enrofloxacina) sono stati utilizzati per l’allestimento di microcosmi sperimentali. Immediatamente dopo il trattamento e a tempi prestabiliti, sono state campionate aliquote di suolo dai vari trattamenti per determinare eventuali differenze nei gruppi microbici presenti (Bacteria e Archaea), presenza di geni di resistenza e per determinare se la concentrazione degli antibiotici diminuisce nel tempo (degradazione).

La analisi dei geni della resistenza agli antibiotici è stata effettuata attraverso l’estrazione del DNA dei microorganismi e amplificazione con qPCR

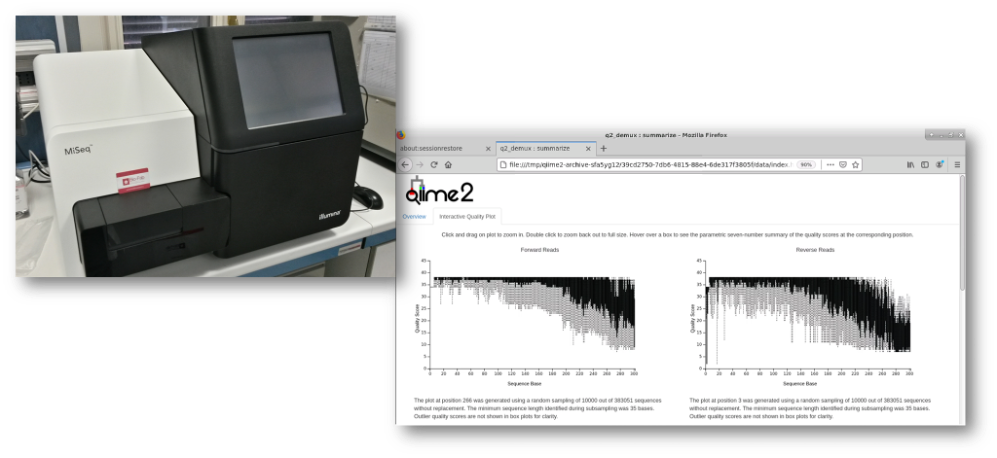
L’analisi della struttura e funzione delle comunità microbiche è stata eseguita tramite sequenziamento di nuova generazione (NGS: Next Generation Sequencing) del gene 16S e analisi bioinformatiche delle sequenze ottenute (Reads).
La determinazione analitica della concentrazione degli antibiotici (sulfametossazolo, ciprofloxacina, enrofloxacina) è stata effettuata tramite metodiche che si avvalgono della cromatografia liquida ad alta prestazione (HPLC), ASE (accelerated solvent extraction) e PLE (pressurized liquid extraction).

L’analisi della struttura della comunità microbica del suolo nei microcosmi è stata analizzata anche con metodi diretti che si avvalgono della microscopia ad epifluorescenza.

Il gruppo di ricerca CNR

Anna Barra Caracciolo
Ruolo
PhD in Ecologia, Coordinatrice del Progetto AZeRO Antibiotici, Dirigente di Ricerca in Risorse naturali ed ecosistemi dell’IRSA-CNR, esperta in Ecologia ed ecologia microbica delle acque e dei suoli.

Paola Grenni
Ruolo
PhD in Scienze Ambientali, Ricercatrice senior IRSA-CNR, esperta in tecniche di ecologia microbica per l’identificazione della struttura e funzione di comunità microbiche naturali di acque e suoli.

Luisa Patrolecco
Ruolo
Chimica ambientale, Ricercatrice senior IRSA-CNR fino al 2019. Dal 2019 è Responsabile di Sede Secondaria di Roma dell’ISP-CNR (Istituto di Scienze Polari). Referente scientifica nel progetto per la valutazione delle dinamiche ambientali degli antibiotici e dei loro metaboliti.

Andrea Visca
Ruolo
PhD in Ingegneria Chimica, Biotecnologo industriale, Assegnista di ricerca presso l’IRSA-CNR, con competenze nell’identificazione e sequenziamento di geni funzionali in comunità microbiche complesse.

Ludovica Rolando
Ruolo
Biologa, dottoranda di ricerca in Ecologia (III anno), esperta in biodegradazione, bioaccessibilità e biodisponibilità dei contaminanti e di chemiotassi batterica.

Gian Luigi Garbini
Ruolo
Dottorando di ricerca in Ecologia, esperto di bioinformatica e di elaborazione dati da sequenziamenti di nuova generazione (NGS).

Livia Mariani
Ruolo
Biologa, Esperta in analisi ecotossicologiche di matrici ambientali (suolo, acqua, sedimento). Esperta in esercizi di inter-calibrazione per validare i metodi ecotossicologici.

Jasmin Rauseo
Ruolo
PhD in Ecologia. Biologa, Ricercatrice ISP-CNR, esperta di processi di biodegradazione di antibiotici, con particolare attenzione ai pattern metabolici e allo sviluppo dell’antibiotico resistenza.

Francesca Spataro
Ruolo
Chimica, PhD in Scienze Chimiche, Ricercatrice ISP-CNR, esperta in monitoraggio di contaminanti emergenti nell’ambiente e messa a punto di tecniche analitiche per la determinazione di antibiotici.

Nicoletta Ademollo
Ruolo
Biologa, PhD in Scienze Polari, Ricercatrice ISP-CNR, esperta in analisi di contaminanti emergenti in matrice biotiche ed abiotiche.

Tanita Pescatore
Ruolo
Dottoranda in Ecologia (III anno), esperta in determinazioni cromatografiche di contaminanti ambientali e loro impatto ecosistemico.

Massimo Ianigro
Ruolo
Informatico, Primo tecnologo del CNR – IRSA, si occupa di infrastrutture e servizi ICT, sicurezza e sviluppo software con tecnologie web. Ha curato la realizzazione del sito,
